바이오 경로 이미지에서 유전자·대사물질 자동 추출하는 AI 프레임워크 개발
기계학습 기반 'EBPI'로 문헌 속 생화학 반응 정보 추출 성공
바이오 경로 이미지에서 유전자·대사물질 자동 추출하는 AI 프레임워크 개발기계학습 기반 'EBPI'로 문헌 속 생화학 반응 정보 추출 성공
- 생명화학공학과 김현욱 교수팀, 대량의 문헌 속 바이오 경로 이미지를 자동으로 분석해, 해당 생화학 반응 정보를 추출하는 기계학습 기반 프레임워크 개발 - 바이오 경로 정보 추출의 자동화를 통해 대규모 데이터 기반 생명공학, 대사공학 및 합성생물학 연구에서 실험 디자인 및 분석 시 핵심적인 역할을 할 것으로 기대
KAIST 생명화학공학과 김현욱 교수 연구팀이 바이오 경로 이미지에서 유전자와 대사물질 정보를 자동 추출하는 EBPI를 개발해, 대규모 문헌 데이터 분석에 활용될 전망이다.
▲(왼쪽부터) KAIST 생명화학공학과 권문수 박사과정, 이준규 박사과정, 김현욱 교수
유전자, 단백질, 대사물질 등 복잡한 정보를 표현하는 바이오 경로 이미지는 중요한 연구 결과를 내포하고 있지만, 이미지 기반 정보 추출에 대해 그동안 충분한 연구가 이뤄지지 않았다. 이에 KAIST 연구진은 바이오 경로 정보를 자동으로 추출할 수 있는 인공지능 프레임워크를 개발했다.
KAIST 생명화학공학과 김현욱 교수 연구팀이 바이오 경로 이미지에서 유전자와 대사물질 정보를 자동으로 추출하는 기계학습 기반의 ‘바이오 경로 정보 추출 프레임워크(이하 EBPI, Extraction of Biological Pathway Information)’를 개발했다고 28일 밝혔다.
연구팀이 개발한 EBPI는 문헌에서 추출한 이미지 속의 화살표와 텍스트를 인식하고, 이를 기반으로 바이오 경로를 편집 가능한 표의 형태로 재구성한다. 객체 감지 모델 등의 기계학습을 사용해 경로 이미지 내 화살표의 위치와 방향을 감지하고, 이미지 속 텍스트를 유전자, 단백질, 대사물질로 분류한다. 그 후 추출된 정보를 통합해 경로 정보를 표 형식으로 제공한다.
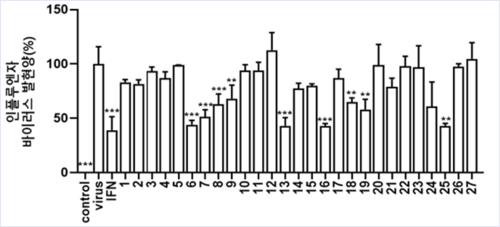
연구팀은 74,853편의 논문에서 추출한 바이오 경로 이미지와 기존 수작업으로 작성된 경로 지도를 비교하며 EBPI의 성능을 검증했다. 그 결과, 높은 정확도로 바이오 경로 정보가 자동으로 추출됐음을 확인했다.
EBPI를 사용해 대표적인 바이오 경로 데이터베이스에 포함되지 않은 생화학 반응 정보를 대량의 문헌 내 바이오 경로 이미지로부터 추출하는 데에도 성공했다.
다양한 산업적 가치를 지닌 대사물질들의 생합성 관련 문헌을 EBPI로 분석한 결과, 문헌에서는 보고가 됐지만, 기존 데이터베이스에서는 누락된 생화학 반응들이 확인된 것이다. 화학산업에서 다양한 응용분야를 갖는 1,4-부탄디올, 2-메틸부티르산, 하이드록시티로솔, 레불린산 및 발레로락탐의 생합성 경로를 예시로 이러한 발견을 제시했다.
연구를 총괄한 김현욱 교수는 “이번 연구에서 개발된 EBPI는 대규모 문헌 데이터 분석에 있어 중요한 도구가 될 것이며 생명공학, 대사공학 및 합성생물학 분야에서 바이오 경로 이미지를 AI로 분석하는 최초의 사례로, 관련 연구의 실험 디자인 및 분석 시 유용하게 활용될 수 있을 것”이라고 밝혔다.
KAIST 생명화학공학과 권문수 박사과정생과 이준규 박사과정생이 공동 제1 저자인 이번 연구는 대사공학 및 합성생물학 분야의 대표적 국제학술지인 대사공학(Metabolic Engineering, JCR 분야 상위 10% 이내)에 11월호에 게재됐다. ※ 논문명 : A machine learning framework for extracting information from biological pathway images in the literature ※ 저자 정보 : 권문수(한국과학기술원, 공동 제1 저자), 이준규(한국과학기술원, 공동 제1 저자), 김현욱(한국과학기술원, 교신저자) 포함 총 3명
한편 이번 연구는 과학기술정보통신부 한국연구재단 및 농촌진흥청의 농업미생물사업단의 지원을 받아 수행됐다.
[그림 1] 바이오 경로 이미지에서 생화학 반응 정보를 자동으로 추출하는 EBPI의 모식도
[그림 2] 감마 하이드록시뷰티르산 대사경로 이미지에 EBPI를 적용하여, 해당 생화학 반응 정보를 추출한 예시
<저작권자 ⓒ 대한건설경제 무단전재 및 재배포 금지>
댓글
|
많이 본 기사
|